NSI Première




import matplotlib.pyplot as plt
import csv
iris=[]
x0,x1,x2=[],[],[]
y0,y1,y2=[],[],[]
esp=[]
with open("iris.csv",'r',encoding='utf-8') as f:
lecteur=csv.reader(f,delimiter=',')
for row in lecteur:
iris.append(row)
for i in range (1,len(iris)):
if iris[i][2]=='0':
x0.append(float(iris[i][0]))
y0.append(float(iris[i][1]))
if iris[i][2]=='1':
x1.append(float(iris[i][0]))
y1.append(float(iris[i][1]))
if iris[i][2]=='2':
x2.append(float(iris[i][0]))
y2.append(float(iris[i][1]))
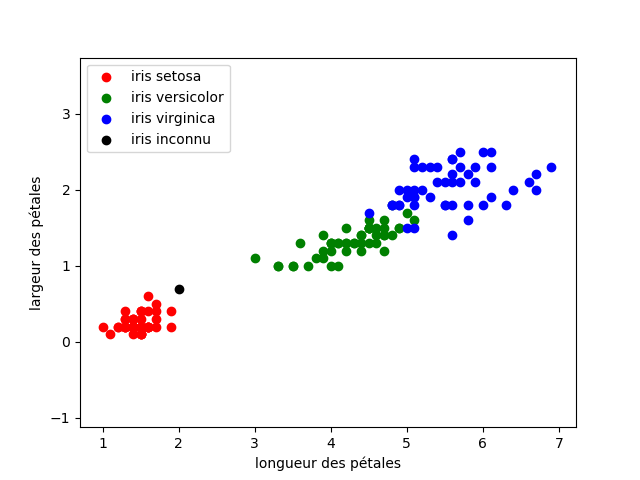
plt.axis('equal')
plt.scatter(x0, y0, color='r', label='iris setosa')
plt.scatter(x1, y1, color='g', label='iris versicolor')
plt.scatter(x2, y2, color='b', label='iris virginica')
plt.legend()
axes = plt.gca()
axes.set_xlabel('longueur des pétales')
axes.set_ylabel('largeur des pétales')
axes.legend()
plt.show()
[['petal_length', 'petal_width', 'species'], [1.4, 0.2, 0], [1.3, 0.2, 0], ...
[['petal_length', 'petal_width', 'species','dist'], [1.4, 0.2, 0, 1.4836666666], [1.3, 0.2, 0, 1.658975975975], ...
recup_donnees(nomDeFichier)->listaffichage_iris(tab:list)distance(x1:float, y1:float, x2:float, y2:float)->floatmini_iris(tab:list)[1.4, 0.2, 0, 1.4836666666]) ainsi que la structure de données "tableau de données et distances" auquel ce sous tableau a été enlevé)kppv_iris(iris:list, larg:float, haut:float,nbvoisins:int)->list:nbvoisins plus proches voisins, recherche l'espèce majoritaire puis ajoute au tableau de données le nouvel iris avec son espèces, et renvoie ce tableau.
import matplotlib.pyplot as plt
import csv
from math import sqrt
def affichage_iris(tab:list):
'''
ne rien toucher à cette fonction
'''
x0,x1,x2=[],[],[]
y0,y1,y2=[],[],[]
esp=[]
for i in range (1,len(tab)):
if tab[i][2]==0:
x0.append(tab[i][0])
y0.append(tab[i][1])
if tab[i][2]==1:
x1.append(tab[i][0])
y1.append(tab[i][1])
if tab[i][2]==2:
x2.append(tab[i][0])
y2.append(tab[i][1])
plt.axis('equal')
plt.scatter(x0, y0, color='r', label='iris setosa')
plt.scatter(x1, y1, color='g', label='iris versicolor')
plt.scatter(x2, y2, color='b', label='iris virginica')
plt.legend()
axes = plt.gca()
axes.set_xlabel('longueur des pétales')
axes.set_ylabel('largeur des pétales')
axes.legend()
plt.show()
def recup_donnees(nomDeFichier)->list:
'''
à partir du nom de fichier, cette fonction récupère les données et les renvoie sous forme de tableau de tableaux
ne rien toucher à cette fonction
'''
iris=[]
with open(nomDeFichier,'r',encoding='utf-8') as f:
lecteur=csv.reader(f,delimiter=',')
for row in lecteur:
iris.append(row)
for i in range (1,len(iris)):
for j in range(2):
iris[i][j]=float(iris[i][j])
iris[i][2]=int(iris[i][j])
return iris
def mini_iris(tab:list):
'''
recherche le minimu et enlève l'élément du tableau de tableaux
renvoie le minimum et le tableau de tableaux modifié
'''
min=100000
indicemin=0
for i in range (len(tab))
if tab[i][3] < min:
min=tab[i][3]
indicemin=i
min=tab.pop(indicemin)
return min,tab
def distance(x1:float,y1:float,x2:float,y2:float)->float
'''
calcule la "distance" entre deux iris
'''
return sqrt((x2-x1)**2+(y2-y1)**2)
def kppv_iris(iris:list, larg:float,haut:float,nbvoisins:int)-> list:
'''
recherche des nbvoisins les plus proches pour déterminer l'espèce de l'iris donc les pétales mesurent larg x haut.
Une fois l'espèce déterminée, le nouvel iris est ajouté au tableau iris qui est renvoyé modifié en fin de fonction.
'''
#copie du tableau de tableaux iris dans le tableau de tableaux iris_kppv
iris_kppv=[[iris[i][j] for j in range(3)]for i in range(1,len(iris))]
#calcul des distances à toutes les autres fleurs et stockage dans le tableau de tableaux iris_kppv
for i in range (0,len(iris_kppv)):
iris_kppv[i].append(distance(larg,haut,iris_kppv[i][0],iris_kppv[i][1]))
#recherche des k voisins les plus proches
voisins=[]
for i in range (nbvoisins):
voisin_proche,iris_kppv=mini_iris(iris_kppv)
voisins.append(voisin_proche)
#recherche de l'espèce majoritaire des k voisins
#le tableau espece va contenir l'effectif de chaque espèce : espece[0]->setosa ; espece[1]->versicolor ; espece[2]->virginica
espece=[0,0,0]
#on parcourt le tableau de tableaux contenant les proches voisins
for i in range (len(voisins)):
if voisins[i][2]==0:
pass # ... supprimer le pass puis compléter la ligne
if voisins[i][2]==1:
pass # ... supprimer le pass puis compléter la ligne
if voisins[i][2]==2:
pass # ... supprimer le pass puis compléter la ligne
#ajout de l'iris inconnu au tableaua de taableaux "iris"
iris.append([larg,haut,espece.index(max(espece))])
return iris
#####################################################
#récupération des données
fleurs=recup_donnees('iris.csv')
#application de l'algorithme des k plus proches voisins
#affichage des données
affichage_iris(fleurs)
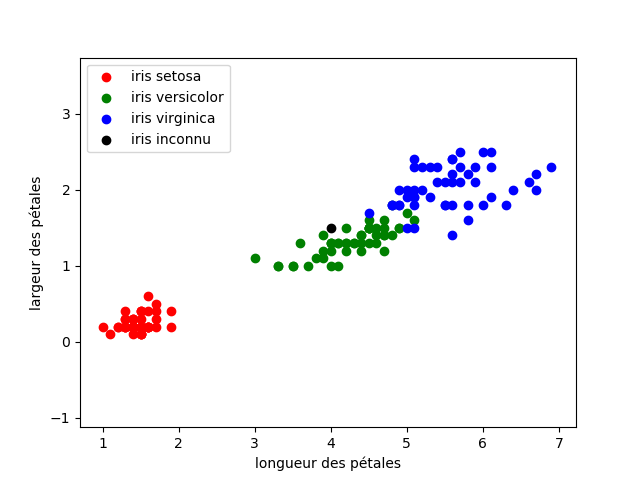
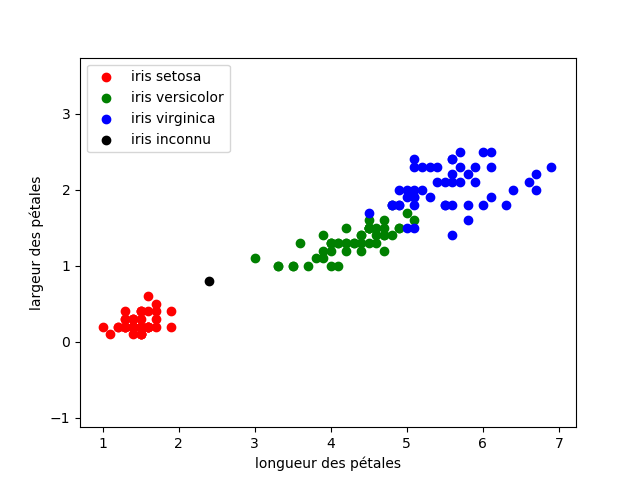
iris.csv. Une erreur de syntaxe apparaît, corrigez làfleursl'iris inconnu dont le pétale mesure 5.5 cm de longueur et 1.95 cm de largeur. On s'appuiera pour cela sur ses 5 plus proches voisins.